Nutzen Sie unser Oligo Analyse Tool zur einfachen Berechnung von Extinktionskoeffizenten.
Der Extinktionskoeffizient bei 260 nm (e260) ist eine eindeutige physikalische Eigenschaft jedes Oligonukleotids. Definiert wird er als die gemessene Absorption bei 260 nm in einer 1 M wässrigen Lösung bei 20°C durch eine optischen Zelle mit einer Durchtrittslänge von 1 cm (Lambert-Beer`sches Gesetz).
Purin-Basen zeigen eine höhere Absorption als Pyrimidin-Basen. Interaktionen zwischen benachbarten Basen, wie auch Modifikationen, die bei 260 nm absorbieren, beeinflussen ebenfalls die optische Absorption. Somit hängt der Extinktionskoeffizient stark von der Oligosequenz und der Basenanordnung ab.
Die ursprüngliche Formel, die Eurofins zur Berechnung der Substanzmenge verwendet, lautete:
nmol = OD x 100 / (1.54 x nA + 0.75 x nC + 1.17 x nG + 0.92 x nT)
(
Jetzt hat Eurofins auch eine neue Formel für die Berechnung des Molaren Extinktionskoeffizient implementiert, die die Bestimmung der Substanzmenge genauer berechnet. Die neue Formel basiert auf der Nearest-Neighbor-Methode die sequenzspezifischen Effekte berücksichtigt:
nmol = OD x 106 / e260
Sowohl der Extinktionskoeffizienten des nächsten Nukleotids sowie der Extinktionskoeffizienten der einzelnen Basen werden in der "Nearest-Neighbor" Formel für die Berechnung des Gesamt-Extinktionskoeffizient eines Oligonukleotids verwendet:
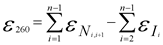
Für Ihre Oligos können Sie in Ihren Einstellungen festlegen, welche Formel zur Berechnung der Oligokonzentration angewendet werden soll.