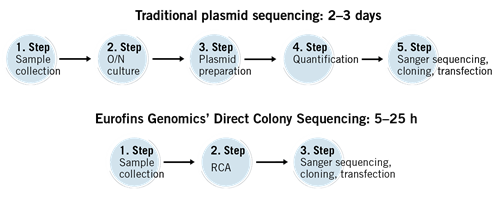
Introduction
You have prepared your DNA construct, cloned it into a vector and transformed E. coli with it. Now it is time to pick and grow the colonies and conduct minipreps in order to extract plasmids to send for sequencing. Confirmation of a construct’s presence and identification of the construct in the vectors is absolutely essential before you continue to work with the plasmids. However, this process is quite laborious and time-consuming, especially when there are a large number of colonies to check.
You can save time by using the Direct Colony Sequencing solution. This Sanger sequencing service in 96-well plates conveniently sequences the plasmids/vectors directly from bacterial colonies without the need for plasmid preparation.
You just need to pick the colonies and directly transfer them to the 96-well plate that comes with the service. The wells of this plate contain buffer to preserve the colonies.
This fast and cost-effective solution can be used to quickly screen your DNA cloning experiments and select the candidate(s) for final confirmation via DNA preparation and sequencing.
What is the basis of Direct Colony Sequencing and why does this sequencing solution work without plasmid preparation?
Rolling circle amplification process
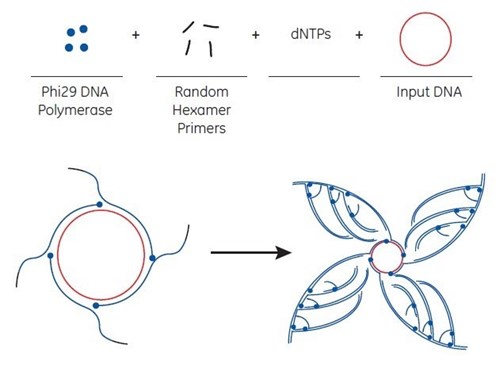
The basis for the Direct Colony Sequencing solution is the rolling circle amplification (RCA) technique that generates multiple copies of circular DNA.
The rolling circle amplification (RCA) method utilises bacteriophage Phi29 DNA polymerase to exponentially amplify single- or double-stranded circular DNA templates. The isothermal amplification method produces microgram quantities of DNA from picogram amounts of starting material in a few hours.
An in vitro amplification of very small amounts of template DNA eliminates the need for overnight cell culture and conventional plasmid or M13 bacteriophage DNA purification. The proofreading activity of the Phi9 DNA polymerase ensures high fidelity DNA replication.
Starting material
The starting material for amplification can be of various sources, such as from a small number of bacterial cells containing a plasmid, isolated plasmids and intact M13 phages to any circular DNA (including ligation reactions).
Bacterial colonies can be picked from agar plates and added directly to the 96-well plate. Alternatively, microlitre quantities of a saturated bacterial culture or a glycerol stock can also serve as starting material.
Process
RCA is performed at 30°C without thermal cycling. Depending on the source of starting material, amplification is completed in 6 to 18 hours. The amplification products are double-stranded concatemers of the circular template with high molecular weight. Concatemers are large DNA molecules that contain many copies of a specific DNA sequence.
For RCA, random hexamer primers anneal to the circular single-strand template DNA at multiple sites. The Phi29 DNA polymerase extends each of these primers. When Phi29 reaches a downstream extended primer, strand displacement synthesis occurs. Here, the original single-stranded DNA is replaced by the newly replicated strand. The displaced strand is then available to be primed by more hexamer primer. The process results in exponential amplification.
Note: when starting with M13 bacteriophage clones, the product is double-stranded DNA and can be sequenced with forward and reverse primers. DNA amplified by the method can be used directly without any purification.
The Direct Colony Sequencing service has been validated primarily for plasmids in E. coli colonies. In principle, the method also works with large constructs (e.g. BACs) and other organisms. Contact us for pilot testing and optimisation. As a starting point, we recommend performing cell lysis or crude DNA extraction and adding 1 to 5 µL of the lysate/extract to the buffer in the 96-well plates.
References
Dean, F. et al., Genome Research 11, 1095–1099 (2001).
Lizardi, P. et al., Nat. Genet. 19, 225–232 (1998).
Estaban, J.A. et al., J. Biol. Chem. 268, 2719–2726 (1993).
Profitieren Sie von größtmöglicher Flexibilität für Ihre Sequenzier-Primer
Dank unserer hauseigenen DNA-Synthese, unserer umfassenden Bioinformatik-und IT-Expertise haben Sie die freie Wahl, wie Sie uns Ihre Sequenzier-Primer zukommen lassen:
- Mehr als 80 Standard Primer stehen Ihnen zur Verfügung
Wählen und verwalten Sie Ihre bevorzugten Standard Primer aus einer Liste von 80 validierten Primern.
- Designen Sie Ihre eigenen Sequenzier-Primer
Verwenden Sie unser Sequenzier Primer Design Tool für Ihre forward und reverse Primer die Sie anschließen einfach bestellen können.
- Bestellen Sie Ihre eigenen Sequenzier-Primer
Bestellen Sie die Synthese benötigter Primer direkt zu Ihrer Sequenzierung. Wir bewahren diese Primer 4 Wochen für Sie auf. In diesem Zeitraum können Sie sie jederzeit bequem wiederverwenden.
- Schicken Sie uns Ihre eigenen Sequenzier-Primer
Sie können uns Ihre Sequenzier-Primer in separaten Tubes gemeinsam mit Ihrer Proben schicken. Verwenden Sie dafür unsere kostenlosen Primer Barcodes.*
Bitte beachten Sie die optimalen Primerbedingungen, Konzentration und Volumen wie unten beschrieben.
Optimale Primer-Bedingungen:
- Primer dürfen weder Phosphorylierung noch Fluoreszenzfarbstoffe enthalten
- Die optimale Primerlänge liegt zwischen 16 und 25 Basen
- Die Schmelztemperatur des Primers sollte 50 - 62°C betragen
- Der GC-Gehalt des Primers sollte bei 35-60% liegen
- Idealerweise sollte ein G oder C am 3’-Ende liegen
- Die Anzahl der 3' Gs oder Cs sollte nicht größer als 2 Gs oder Cs sein
- Wenn möglich, vermeiden Sie >3 identische Basen hintereinander in der Sequenz
Primer-Konzentration und -Volumen:
- Pro Sequenzierreaktion werden exakt 10 pmol/µl Primer-Konzentration benötigt
- Jeder Primer muss ein Gesamtvolumen von 15 µl (in bi-destilliertem Wasser oder 5mM Tris-HCl) haben; für jede weitere Sequenzierreaktion sind 5 µl Primer-Volumen erforderlich
- Die Konzentration von Primern mit Wobbles muss nach folgender Formel berechnet werden: nX x ConcPrimer