Was Sie von unseren NGSgrade Oligos 2.0 erwarten können
- Typische Kreuzkontaminationsrate von 0,01 % bis 0,05 %, bestätigt durch unser NGS-Labor (siehe Kreuzkontaminationsrate)
- Verfügbare Mindestmengen von 4 nmol, 10 nmol und 25 nmol
- DNA-Sequenzlänge von 15–120 Basen
- Verfügbare Modifikationen sind PTO-Bindungen, 2’-Desoxyinosin, 2’-Desoxyuridin und 5’-Phosphat
- Die Qualität wird durch OD-Messung und ESI-MS (Elektrospray-Ionisations-Massenspektrometrie) sichergestellt.
- Ein Online-Qualitätsbericht, einschließlich der ESI-MS-Spektren, kann bestellt werden
- Auswählbare Lösungsmittel sind bidestilliertes Wasser, TE-Puffer (10 mM Tris, 1 mM EDTA; pH = 8) oder Low EDTA Puffer (10 mM Tris, 0,1 mM EDTA, pH = 8)
- Schnelle Lieferzeiten ab 8 Arbeitstagen
Zur Lieferung in Tubes:
- Oligos werden in 2ml Tubes mit Schraubverschluss entweder lyophilisiert oder in flüssiger Form in der ausgewählten Konzentration geliefert
Zur Lieferung in Platten:
- Oligos werden in der ausgewählten 96-Well-Platte in flüssiger Form in der definierten Konzentration geliefert
- Es sind mindestens 20 Oligos pro 96-Well-Platte erforderlich
Die Kreuzkontaminationsrate von 0,01 bis 0,05 % wurde durch Sequenzierung repräsentativer Sets von Unique Dual Index (UDI)-Primer-Paaren unter Verwendung von Illumina-Instrumenten in der europäischen NGS-Sequenzierungsanlage von Eurofins Genomics bestimmt. Die angegebene Rate stellt den typischen Gesamtprozentsatz der Lese-Paare dar, die nicht den erwarteten UDI-Kombinationen für einen Satz von 96 UDI-Primer-Paaren entsprechen. Die entsprechende Anwendungsnotiz zeigt eine Kreuzkontaminationsrate von 0,0014 % für einen Satz von 14 UDI-Primer-Paaren.
Bitte beachten Sie, dass die Gesamtkreuzkontaminationsrate (Prozentsatz der falsch zugeordneten Lese-Paare) von der Gesamtanzahl der analysierten UDI-Primer-Paare parallel beeinflusst wird. Je mehr UDI-Primer analysiert werden, desto höher kann die Gesamtkreuzkontamination sein, da mehr unerwartete Indexkombinationen zugeordnet werden können.
In allen Fällen wäre die tatsächliche Kreuzkontamination der Primer sogar noch geringer, wie oben angegeben. Dies liegt daran, dass die Zuordnung von Lesevorgängen zu anderen unerwarteten UDI-Primer-Paaren eine Folge von Primer-Kreuzkontamination und Index-Hopping während der Sequenzierung ist. Eine Unterscheidung zwischen diesen Ursachen ist in diesem Analyse-Setup nicht möglich.
Bitte beachten Sie, dass Eurofins Genomics die Kreuzkontaminationsrate für jeden einzelnen Satz von produzierten und versandten NGS-Oligonukleotiden nicht bestimmt.
| |
Gesamte Kreuzkontaminationsrate
pro UDI-Satz (% Gesamtlesepaare, die nicht den erwarteten UDI-Kombinationen entsprechen) |
Die niedrigste gemessene Kreuzkontamination pro UDI-Kombination |
Referenz |
| Eurofins Genomics NGSgrade Oligos 2.0 |
Typisch 0.01 - 0.05% |
0.00446% |
Set of 96 UDI Primer-Paaren |
| Eurofins Genomics NGSgrade Oligos 2.0 |
0.0014%
(siehe Application Note) |
0.00045% |
Set of 14 UDI Primer-Paaren |
| Mitbewerber |
0.0208%
(siehe Application Note) |
0.00767% |
Set of 14 UDI Primer-Paaren |
Produktdaten
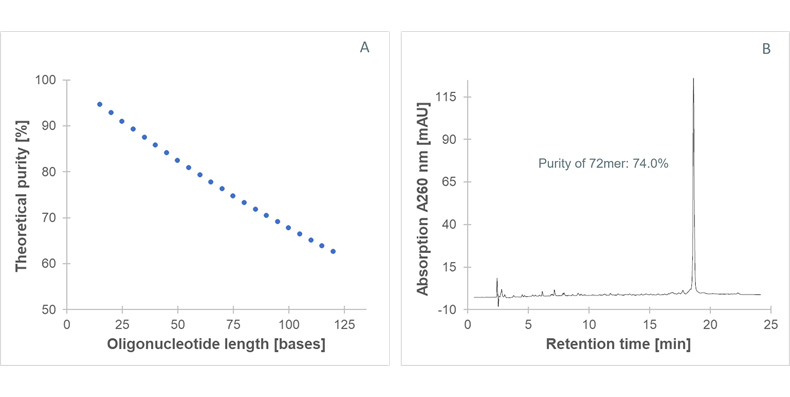
Eurofins Genomics verwendet ein neues und proprietäres Synthesegerät zur Herstellung der hochqualitativen NGSgrade 2.0 Oligonukleotide. Dieses Gerät ermöglicht eine unvergleichliche Kopplungseffizienz, und damit zu einem sehr hohen Prozentsatz an Vollängen-Oligonukleotiden.
Adapter-Ligation-Oligos für die NGS-Amplicon-Sequenzierung auf NovaSeq
Hochwertige Adapter-Oligos zur Verbesserung des Multiplexings mit Proben-Tagging durch NGS auf Illumina. Sie wurden in unserem firmeneigenen NextGen-Sequenzierungslabor entwickelt und validiert:
- 192 individuell gestaltete Tags stehen zur Kennzeichnung Ihrer Proben zur Verfügung
- Jeder Tag besteht aus 10 Nukleotiden, die zu Ihrem templatespezifischen Forward-Primer* hinzugefügt werden*.
- Die Tags sind mit einem minimalen Editierabstand von 4 konzipiert, um Fehlzuweisungen durch Sequenzierfehler bestmöglich zu vermeiden
- Optimiert für Probenpools mit geringer und hoher Probenanzahl
- Alle NGSgrade Oligos durchlaufen spezifische, kontaminationsfreie Prozesse, um höchste Reinheit und Leistung zu gewährleisten
Bitte verwenden Sie unsere Excel-Vorlage die auf der NGSgrade 2.0 Oligo-Bestellseite bereitgestellt wird. Diese ist speziell für die Gestaltung Ihrer Amplicon-Adapter-Ligation-Oligos kodiert.
Die entwickelten Primer können sowohl mit unserem NGSelect Amplicon Adapter Ligation-Produkt als auch mit Ihrem eigenen Sequenzierprotokoll verwendet werden.
2nd PCR Primer für NGS-Amplikon-Sequenzierung auf MiSeq
Die Amplicon 2nd PCR Primer sind speziell entwickelt, um das Pooling von Proben für die NGS-Sequenzierung auf Illumina-Plattformen zu ermöglichen. Sie wurden von unserem hauseigenen NextGen-Sequenzierungslabor entworfen und validiert.
Bei der NGS-Amplikon-Sequenzierung auf MiSeq werden Primer mit universellen Adaptern für die erste PCR benötigt.
Zum Komfort unserer Kunden fügt Eurofins Genomics diese Adapter* automatisch zu Ihren zielsequenzspezifischen Primern hinzu. Dieser automatisierte Prozess während der Online-Bestellung stellt sicher, dass Ihre Primer korrekt, validiert und optimiert sind. Bitte verwenden Sie hierfür unsere Excel-Vorlage, die auf der Bestellseite für NGSgrade 2.0 Oligos verfügbar ist.
Adaptersequenzen:
- Forward-Primer-Sequenz: ACACTCTTTCCCTACACGACGCTCTTCCGATCT
- Reverse-Primer-Sequenz: GACTGGAGTTCAGACGTGTGCTCTTCCGATCT
Die entworfenen Primer sind mit unserem NGSelect Amplicon MiSeq-Service kompatibel.
Was Sie von unseren NGSgrade Oligos erwarten können
Optimierte und verifizierte Produktspezifikationen:
- Garantierte Reinheit von >80% durch HPLC Reinigung
- Spezielle kreuzkontaminationsfreie Prozessabläufe nach Synthese
- Strenge Qualitätskriterien, überprüft durch MALDI-TOF MS
- Synthesemaßstäbe von 0.01 bis 1.0 µmol
- Wählbares Lieferformat: Lyophilisiert oder konzentrationseingestellt
- Lieferzeiten:
- 5-7 Werktage für bis zu 60 mere
- 10 Werktage für > 61 mere
Minimum Ausbeuten (OD) pro Scale:
|
Synthesis scale
|
0.01 µmol
|
0.05 µmol
|
0.2 µmol
|
1.0 µmol
|
HPLC
|
Min. Ausbeute [OD] |
1 |
2 |
4 |
10 |
Zusätzliche kostenlose Online Funktionen
- Fügen Sie eine persönliche Note zu ihrer Oligo resp. Platte
- Berechnen Sie die Komplementärfolge
- Auswahl unterschiedlicher Tube-Etiketten
- Verfolgen Sie ihren Produktions- und Lieferstatus Online
- Sehen Sie sich Ihren Bestellvorlauf unter "Meine Bestellungen" an