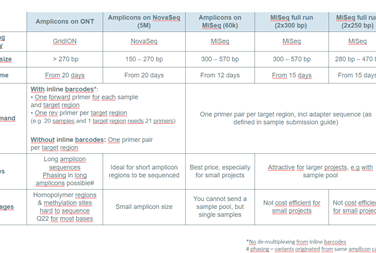
Die folgende Tabelle soll helfen die Richtige Technology für die Amplikonsequenzierung auszuwählen.
Bitte klicken Sie auf das Bild um es zu vergrößern:
Amplikon-Sequenzierung mittels Adaptor Ligation
Der Adaptor Ligation Ansatz kann für die Sequenzierung kurzer Amplikons mit der Illumina Technologie verwendet werden.
Bei der Illumina Sequenzierung von Amplikons können Sie eine sehr hohe Coverage erzielen, und dadurch somatische Mutationen in komplexen Proben identifizieren. Proprietäre Protokolle und jahrelange NGS Expertise machen eine hohe Datenqualität mit außerordentlich kurzen Lieferzeiten möglich.
Besonderheiten von NGSelect Amplikons Adaptor Ligation
- Hochsensitive NGS-Arbeitsabläufe mit reduziertem Bias
- Flexible Datenpakete ab 5 M Read Paaren
- Multiplexing von bis zu 192 Illumina Proben
- Wenn Sie mehrere Proben multiplexen wollen: Eurofins Primer Design um Ihnen Zeit zu sparen, und Ihnen Best Practice Multiplexing anzubieten
Ausgangsmaterial
|
Bibliothekstyp
|
Ausgangsmaterial
|
Adapter-Ligation
|
100 - 200 ng PCR Produkte / Amplikons (bis zu 100 µl, Konzentration > 1 ng/µl
Für die Sequenzierung mit 5M Read-Pairs und 2 x 150 bp Leselänge sollte die Größe der Amplikons idealerweise zwischen 150 bp und 270 bp
|
Sequenzierpakete
- Garantiert 5M Read Paare (10 Millionen reads) pro Paket, 2 x 150 bp
Bioinformaticsche Analyse
- Zusammenführung überlappender Paired-End-Reads (Merging)
- Sortieren der Sequenzen nach Tags
- vom Kunden designte Tags
- von Eurofins Genomcis designte Tags
Lieferzeit
Ab knapp zwei Wochen; von individuellen Projektzielen abhängig
Wir erledigen das Barcoding und Probenpooling für Sie
Die 2. PCR von NGSelect Amplikon beginnt mit PCR-Proben, die Sie in Ihrem Labor mit Universaladaptern (TruSeq Adapter) vorbereiten. Wir führen die Indexierung Ihrer Proben durch und dann erfolgt die Sequenzierung auf dem MiSeq mit entweder 2 x 300 bp oder 2 x 250 bp, je nach Ihren Anforderungen.
Besonderheiten von NGSelect Amplicon
- Sie brauchen selbst kein Barcoding durchzuführen. Sie brauchen daher auch nur ein Primerpaar pro Zielregion
- Kostengünstiges Outsourcing der verbleibenden Schritte
- Einfaches Primerdesign und Primerbestellung über Eurofins
Ausgangsmaterial
|
Bibliothekstyp
|
Ausgangsmaterial
|
Illumina MiSeq
|
25 µl aufgereinigtes Amplikon Produkt, bevorzugt in EB Puffer (PCR Produkt)1
Die Größe der Amplikons (ohne die "universal adaptors) sollte im Bereich von 280 bp bis 570 bp liegen.
Eine Qualitätskontrolle muss mittels eines Agarose Gels erfolgen (= deutliche Bande(n) der erwarteten Größe unter der Verwendung von 5 µl PCR Produkt).
Die Proben in 1.5ml "safe-lock" Reaktionsröhrchen (Tubes) geschickt werden.
|
Sequencing possibilities
Standard:
- Garantiert 60 k Read Paare pro Amplikon
- 2 x 300 bp Read Modus
Custom:
- Ganze MiSeq flow cell (2 x 300 bp oder 2 x 250 bp)
Sie haben eine große Anzahl von Proben, die Sie mit diesem Workflow analysieren möchten? In diesen Fällen können wir ein maßgeschneidertes Projekt mit vollständigen MiSeq-Läufen anbieten. Bitte kontaktieren Sie uns für eine Projektbesprechung.
Sequenzierung großer und/oder repetitiver Amplicons.
Produktbeschreibung
Bibliotheksherstellung
- ONT-Adapterligation (Multiplexing hängt von der Amplicon-Größe und der bevorzugten Abdeckung ab, max. 96 Proben pro Flow Cell)
Sequenzierung
- Technologie: Oxford Nanopore
- Ausgabeleselänge: Gleich der Fragmentlänge (kann bis zu mehreren kbp betragen)
- Die Durchsatzleistung hängt von der Qualität der eingereichten Probe ab
- Ca. 7 Gb pro Flow Cell (abhängig von der DNA-Qualität und -Länge)
Lieferumfang
- Rohdaten der Nanopore-Amplicons (FastQ)
- BioIT auf Anfrage (z. B. Variantenanalyse VCF-Datei)
Erforderliche Eingabe (abhängig vom Multiplexing)
- OD 260/280: 1,8; OD 260/230: 2,0-2,2
- Max. Amplicon-Größe: 20 kb
- Für 1 Probe:
- Optimal: >25ng/μl in 60 μl
- Für 2-48 Proben:
- Der Größenbereich verschiedener Amplicons sollte ähnlich sein (+/-10 %)
- Optimal: >25ng/μl in 30 μl
So einfach wie noch nie
Für die Illumina Sequenzierung von Amplikons mit dem Adaptor Ligation Protokoll, können Sie entweder Amplikons ohne jegliche Adaptoren vorbereiten, oder unsere Barcoding (=Tagging) Strategie nutzen. Der Tagging Ansatz reduziert die Sequenzierkosten Ihres Projektes, insbesondere dann, wenn Sie Projekte mit mehr als ein paar einzelnen Proben planen.
Option ohne Tagging:
- Definieren Sie den Zielbereich, den Sie amplifizieren möchten, und konzipieren Sie Primer für die Amplifikation dieses Bereiches. Die zu amplifizierenden Region soll zwischen 150 und 270 bp lang sein.
- Bestellen Sie die Zielregion-spezifischen Primer als NGSgrade oligo primers und generieren Sie die Amplikons für alle DNA-Templates (=Proben)
- Überprüfen Sie den Amplifikationserfolg auf einem Gel und reinigen Sie die Amplikons auf
- Da Sie kein Barcoding der einzelnen Proben verwendet haben müssen Sie mindestens 1 Datenpaket pro Amplikon bestellen, um die Reads nach der Sequenzierung einer Probe zuordnen zu können.
Option mit Tagging:
- Definieren Sie den Zielbereich, den Sie amplifizieren möchten, und konzipieren Sie Primer für die Amplifikation dieses Bereiches. Die zu amplifizierenden Region soll zwischen 150 und 270 bp lang sein.
- Laden Sie Ihre zielspezifischen Sequenzen auf unserer Oligo-Bestellseite hoch und überlassen Sie Eurofins das automatische Primerdesign.
- Bestellen Sie Primer und bereiten Sie Amplikons mit den dafür vorgesehenen Primern vor.
- Überprüfen Sie den Amplifikationserfolg auf einem Gel und reinigen und quantifizieren Sie jedes Amplikon für das equimolare Pooling
- Die Verwendung von getaggten Primern ermöglicht die Multiplex-Sequenzierung mehrerer gepoolter Amplifikate in einem einzigen Datenpaket.
So einfach wie noch nie
- Definieren Sie den Zielbereich, den Sie amplifizieren möchten, und konzipieren Sie Primer für die Amplifikation dieses Bereichs. Die zu amplifizierende Region soll zwischen 280 und 570 bp lang sein. bp.
- Laden Sie Ihre zielspezifische Sequenz auf unserer Oligo-Bestellseite hoch und überlassen Sie Eurofins das automatische Primerdesign.
- Bestellen Sie die Primer und führen Sie die Amplifikation mit den vorgesehenen Primern durch.
- Überprüfen Sie den Amplifikationserfolg auf einem Gel und senden Sie uns Ihre aufgereinigten Amplikons zu.
Sie haben noch weitere Fragen? Bitte kontaktieren Sie uns.